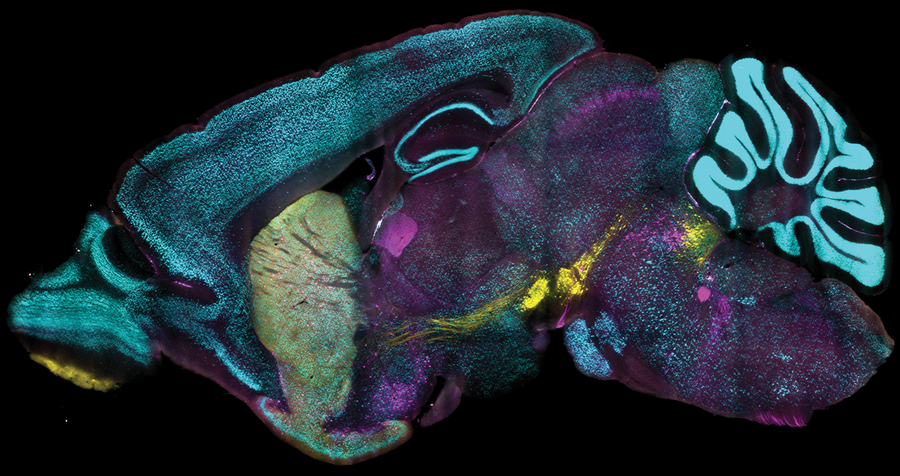
El avance en el procesamiento de tejidos puede etiquetar proteínas a nivel de células individuales en muestras grandes con tanta rapidez y uniformidad como en células individuales disociadas
Por: David Orenstein |MIT | Instituto Picower para el Aprendizaje y la Memoria
Una nueva tecnología desarrollada en el MIT permite a los científicos etiquetar proteínas en millones de células individuales en tejidos 3D completamente intactos con una velocidad, uniformidad y versatilidad sin precedentes. Utilizando la tecnología, el equipo pudo etiquetar en gran cantidad muestras de tejido de gran tamaño en un solo día. En su nuevo estudio en Nature Biotechnology , también demuestran que la capacidad de etiquetar proteínas con anticuerpos a nivel de célula única en muestras de tejido de gran tamaño puede revelar información que otros métodos de etiquetado ampliamente utilizados no han logrado obtener.
El análisis de las proteínas que las células producen es un elemento básico de los estudios en biología, neurociencia y campos relacionados, porque las proteínas que una célula está expresando en un momento dado pueden reflejar las funciones que la célula está tratando de realizar o su respuesta a las circunstancias, como una enfermedad o un tratamiento. A pesar de los avances en las tecnologías de microscopía y etiquetado, que han permitido innumerables descubrimientos, los científicos aún carecen de una forma fiable y práctica de rastrear la expresión de proteínas a nivel de millones de células individuales densamente compactas en tejidos completos, intactos y tridimensionales. A menudo se limita a secciones delgadas de tejido bajo portaobjetos, por lo que los científicos no han tenido las herramientas para apreciar en profundidad la expresión de proteínas celulares en los sistemas completos y conectados en los que se produce.
“Convencionalmente, investigar las moléculas dentro de las células requiere disociar el tejido en células individuales o cortarlo en secciones delgadas, ya que la luz y los químicos necesarios para el análisis no pueden penetrar profundamente en los tejidos. Nuestro laboratorio desarrolló tecnologías como CLARITY y SHIELD , que permiten la investigación de órganos completos al hacerlos transparentes, pero ahora necesitábamos una forma de etiquetar químicamente órganos completos para obtener información científica útil”, dice el autor principal del estudio Kwanghun Chung , profesor asociado en el Instituto Picower para el Aprendizaje y la Memoria, los departamentos de Ingeniería Química y Ciencias Cognitivas y Cerebrales, y el Instituto de Ingeniería Médica y Ciencia del MIT. “Si las células dentro de un tejido no se procesan de manera uniforme, no se pueden comparar cuantitativamente. En el etiquetado de proteínas convencional, estas moléculas pueden tardar semanas en difundirse en órganos intactos, lo que hace que el procesamiento químico uniforme de los tejidos a escala de órganos sea prácticamente imposible y extremadamente lento”.
Vídeo sobre el etiquetado de proteínas con CuRVE y eFLASH : The Picower Institute
El nuevo método, denominado “CuRVE”, representa un avance importante (que se ha estado gestando durante años) hacia ese objetivo, ya que demuestra un método fundamentalmente nuevo para procesar de manera uniforme tejidos grandes y densos. En el estudio, los investigadores explican cómo superaron las barreras técnicas mediante una implementación de CuRVE denominada “eFLASH” y brindan abundantes demostraciones gráficas de la tecnología, incluido el modo en que generó nuevos conocimientos neurocientíficos.
“Este es un gran avance, especialmente en términos del desempeño real de la tecnología”, dice el coautor principal Dae Hee Yun PhD ’24, un estudiante de posgrado del MIT que ahora es ingeniero de aplicaciones senior en LifeCanvas Technologies, una empresa emergente que Chung fundó para difundir las herramientas que inventa su laboratorio. El otro autor principal del artículo es Young-Gyun Park, un ex posdoctorado del MIT que ahora es profesor asistente en KAIST en Corea del Sur.
Química inteligente
La razón fundamental por la que es difícil etiquetar de manera uniforme muestras de tejido tridimensionales de gran tamaño es que los anticuerpos se infiltran en el tejido muy lentamente, pero se unen rápidamente a las proteínas a las que se dirigen. El efecto práctico de este desajuste de velocidad es que, simplemente sumergiendo un cerebro en un baño de anticuerpos, las proteínas estarán muy bien etiquetadas en el borde exterior del tejido, pero prácticamente ninguno de los anticuerpos encontrará células y proteínas en el interior.
Para mejorar el etiquetado, el equipo ideó una forma (la esencia conceptual de CuRVE) de resolver el desajuste de velocidad. La estrategia consistía en controlar continuamente el ritmo de unión de los anticuerpos y, al mismo tiempo, acelerar la penetración de los mismos en todo el tejido. Para averiguar cómo podría funcionar esto y optimizar el enfoque, crearon y ejecutaron una sofisticada simulación computacional que les permitió probar diferentes configuraciones y parámetros, incluidas diferentes tasas de unión y densidades y composiciones de tejido.
Luego se propusieron implementar su enfoque en tejidos reales. Su punto de partida fue una tecnología anterior, llamada » SWITCH «, en la que el laboratorio de Chung ideó una forma de desactivar temporalmente la unión de los anticuerpos, dejando que los anticuerpos permearan el tejido y luego volviendo a activar la unión. A pesar de que funcionó bien, dice Yun, el equipo se dio cuenta de que podría haber mejoras sustanciales si la velocidad de unión de los anticuerpos pudiera controlarse constantemente, pero los productos químicos utilizados en SWITCH eran demasiado agresivos para un tratamiento tan continuo. Entonces, el equipo examinó una biblioteca de productos químicos similares para encontrar uno que pudiera reducir de manera más sutil y continua la velocidad de unión de los anticuerpos. Descubrieron que el ácido desoxicólico era un candidato ideal. Usando ese producto químico, el equipo no solo podía modular la unión de los anticuerpos variando la concentración del producto químico, sino también variando el pH (o acidez) del baño de etiquetado.
Mientras tanto, para acelerar el movimiento de los anticuerpos a través de los tejidos, el equipo utilizó otra tecnología anterior inventada en el laboratorio de Chung: el electrotransporte estocástico, que acelera la dispersión de los anticuerpos a través de los tejidos mediante la aplicación de campos eléctricos.
La implementación de este sistema eFLASH de dispersión acelerada con velocidad de unión continuamente modificable produjo la amplia variedad de éxitos de etiquetado demostrados en el artículo. En total, el equipo informó haber utilizado más de 60 anticuerpos diferentes para etiquetar proteínas en células en muestras de tejido de gran tamaño.
Cabe destacar que cada una de estas muestras fue etiquetada en un día, una velocidad “ultrarrápida” para órganos enteros e intactos, dicen los autores. Además, las diferentes preparaciones no requirieron nuevos pasos de optimización.
Visualizaciones valiosas
Entre las formas en que el equipo puso a prueba eFLASH se encontraba la comparación de su etiquetado con otro método de uso frecuente: la ingeniería genética de células para que emitan fluorescencia cuando se está transcribiendo el gen de una proteína de interés. El método genético no requiere la dispersión de anticuerpos por todo el tejido, pero puede ser propenso a discrepancias porque informar sobre la transcripción genética y la producción real de proteínas no es exactamente lo mismo. Yun añadió que, si bien el etiquetado con anticuerpos informa de manera fiable e inmediata sobre la presencia de una proteína objetivo, el método genético puede ser mucho menos inmediato y persistente, y seguir emitiendo fluorescencia incluso cuando la proteína real ya no está presente.
En el estudio, el equipo empleó ambos tipos de etiquetado simultáneamente en las muestras. Al visualizar las etiquetas de esa manera, vieron muchos ejemplos en los que el etiquetado de anticuerpos y el etiquetado genético diferían ampliamente. En algunas áreas de cerebros de ratones, descubrieron que dos tercios de las neuronas que expresaban PV (una proteína prominente en ciertas neuronas inhibidoras) según el etiquetado de anticuerpos, no mostraban ninguna fluorescencia de base genética. En otro ejemplo, solo una pequeña fracción de células que informaron la expresión mediante el método genético de una proteína llamada ChAT también lo informaron mediante el etiquetado de anticuerpos. En otras palabras, hubo casos en los que el etiquetado genético informó tanto de forma muy insuficiente como excesiva la expresión de la proteína en comparación con el etiquetado de anticuerpos.
Los investigadores no pretenden cuestionar el valor claro de utilizar los métodos de informe genético, sino que sugieren que también el uso del etiquetado de anticuerpos en todo el órgano, como permite eFLASH, puede ayudar a poner esos datos en un contexto más rico y completo. «Nuestro descubrimiento de una gran pérdida regionalizada de neuronas inmunorreactivas al PV en ratones adultos sanos y con una alta variabilidad individual enfatiza la importancia de la fenotipificación holística e imparcial», escriben los autores.
O, como dice Yun, los dos tipos diferentes de etiquetado son “dos herramientas diferentes para el trabajo”.
Además de Yun, Park y Chung, los otros autores del artículo son Jae Hun Cho, Lee Kamentsky, Nicholas Evans, Nicholas DiNapoli, Katherine Xie, Seo Woo Choi, Alexandre Albanese, Yuxuan Tian, Chang Ho Sohn, Qiangge Zhang, Minyoung Kim, Justin Swaney, Webster Guan, Juhyuk Park, Gabi Drummond, Heejin Choi, Luzdary Ruelas y Guoping Feng.