Las técnicas de arte digital ahora pueden diseñar biomoléculas de trabajo personalizadas bajo demanda
Por: ewen callaway
(nature)- David Juergens, químico computacional de la Universidad de Washington (UW) en Seattle, está a punto de diseñar una proteína que, en más de 3 mil millones de años de retoques, la evolución nunca ha producido.
En una videollamada, Juergens abre una versión basada en la nube de una herramienta de inteligencia artificial (IA) que ayudó a desarrollar, llamada RFdiffusion. Esta red neuronal, y otras similares, están ayudando a llevar la creación de proteínas personalizadas, hasta hace poco una búsqueda altamente técnica y, a menudo, sin éxito, a la ciencia convencional.
Estas proteínas podrían formar la base de vacunas, terapias y biomateriales. “Ha sido un momento completamente transformador”, dice Gevorg Grigoryan, cofundador y director técnico de Generate Biomedicines en Somerville, Massachusetts, una empresa de biotecnología que aplica el diseño de proteínas al desarrollo de fármacos.
Las herramientas están inspiradas en el software de inteligencia artificial que sintetiza imágenes realistas, como el software Midjourney que, este año, se usó para producir una imagen viral del Papa Francisco con una chaqueta blanca acolchada de diseñador. Los investigadores descubrieron que un enfoque conceptual similar puede generar formas de proteínas realistas según los criterios que especifican los diseñadores, lo que significa, por ejemplo, que es posible elaborar rápidamente nuevas proteínas que deberían unirse estrechamente a otra biomolécula. Y los primeros experimentos muestran que cuando los investigadores fabrican estas proteínas, una fracción útil funciona como sugiere el software.
Las herramientas han revolucionado el proceso de diseño de proteínas en el último año, dicen los investigadores. “Es una explosión de capacidades”, dice Mohammed AlQuraishi, biólogo computacional de la Universidad de Columbia en la ciudad de Nueva York, cuyo equipo ha desarrollado una de esas herramientas para el diseño de proteínas. “Ahora puede crear diseños que tienen cualidades buscadas”.
“Estás construyendo una estructura de proteína personalizada para un problema”, dice David Baker, un biofísico computacional de la UW cuyo grupo, que incluye a Juergens, desarrolló RFdiffusion. El equipo lanzó el software en marzo de 2023 , y esta semana aparece un artículo que describe la red neuronal en Nature 1 . (Se lanzó una versión preliminar a fines de 2022, aproximadamente al mismo tiempo que varios otros equipos, incluidos AlQuraishi’s 2 y Grigoryan’s 3 , informaron redes neuronales similares).
Por primera vez, los diseñadores de proteínas ahora tienen los tipos de herramientas robustas y reproducibles en torno a las cuales se puede crear una nueva industria, agrega Grigoryan. “El próximo desafío es, ¿qué haces con él?”
Grandes diseños
Juergens ingresa algunas especificaciones para la proteína que desea en un formulario web que se asemeja a una calculadora de impuestos en línea. Debe tener 100 aminoácidos de largo y formar un complejo simétrico de dos proteínas llamado homodímero. Muchos receptores celulares adoptan esta configuración, y un nuevo homodímero podría ser una molécula de señalización celular sintética, dice Joe Watson, un bioquímico computacional de la UW que co-desarrolló RFdiffusion, y también está en la videollamada. Pero el diseño de esta mañana no pretende hacer nada más que parecerse a una proteína realista.
Los investigadores han luchado durante décadas para construir nuevas proteínas. Al principio, intentaron improvisar partes útiles de proteínas existentes, como una bolsa de una enzima en la que se cataliza una reacción química. Este enfoque se basó en la comprensión de cómo se pliegan y funcionan las proteínas, así como en la intuición y mucho ensayo y error. En ocasiones, los científicos examinaron miles de diseños para identificar uno que funcionara como se esperaba.
Un momento de iluminación llegó con AlphaFold (desarrollado por la firma de IA con sede en Londres DeepMind, ahora Google DeepMind) y otros modelos basados en IA que podían predecir con precisión estructuras de proteínas a partir de secuencias de aminoácidos, dice Baker. Los diseñadores se dieron cuenta de que estas redes neuronales, entrenadas en secuencias y estructuras de proteínas reales, también podrían ayudar a crear proteínas desde cero.
En los últimos años, el equipo de Baker y otros en el campo han lanzado una gran cantidad de herramientas de diseño de proteínas basadas en IA . Un enfoque que utilizan estas herramientas, llamado alucinación, implica la creación de una cadena aleatoria de aminoácidos que luego es optimizada por AlphaFold, o una herramienta similar llamada RoseTTAFold, hasta que se parece a algo que la red neuronal sugiere que es probable que se pliegue en una estructura específica. Otro, llamado inpainting, toma un fragmento específico de una secuencia o estructura de proteína y construye el resto de la molécula a su alrededor usando RoseTTAFold.
Pero estas herramientas están lejos de ser perfectas. Los experimentos tendieron a mostrar que las estructuras diseñadas por métodos de alucinación no siempre formaban proteínas bien plegadas cuando se fabricaban en el laboratorio y terminaban como suciedad en el fondo de un tubo de ensayo, por ejemplo. Los métodos de alucinación también tuvieron problemas para hacer cualquier cosa menos proteínas pequeñas (aunque otros investigadores mostraron, en una preimpresión de febrero, cómo la técnica podría usarse para diseñar moléculas más largas 4 ). Repintar también hizo un mal trabajo en la formación de proteínas cuando se les dieron fragmentos más cortos. Incluso cuando el enfoque produjo una estructura de proteína teórica, no pudo encontrar diversas soluciones a un problema que aumentaría las probabilidades de éxito.
Ahí es donde entran en juego RFdiffusion y otras IA similares de diseño de proteínas, lanzadas en los últimos meses. Se basan en los mismos principios que las redes neuronales que generan imágenes realistas, como Stable Diffusion, DALL-E y Midjourney. Estas redes de ‘difusión’ se entrenan con datos, ya sean imágenes o estructuras de proteínas, que luego se vuelven progresivamente más ruidosas, y finalmente no se parecen a la imagen o estructura inicial. Luego, la red aprende a «eliminar el ruido» de los datos, realizando la tarea a la inversa.
Las redes como RFdiffusion se entrenan en decenas de miles de estructuras de proteínas reales almacenadas en un repositorio llamado Protein Data Bank (PDB). Cuando la red produce una nueva proteína, comienza con un ruido total: una variedad aleatoria de aminoácidos. “Estás preguntando cuál es la proteína que originó el ruido”, explica Watson. Después de rondas de eliminación de ruido, produce algo parecido a una proteína real, pero nueva.
Cuando el equipo de Baker probó la difusión de RF sin proporcionar ninguna guía, excepto la longitud de la proteína, la red generó proteínas diversas y de aspecto realista, diferentes de todo lo que había sido entrenado en el PDB.
Pero los investigadores también pueden dirigir el programa para que produzca proteínas de acuerdo con restricciones de diseño específicas durante el proceso de eliminación de ruido, un proceso llamado condicionamiento.
Por ejemplo, el equipo de Baker condicionó la difusión de RF para producir proteínas que incluyan un pliegue específico o que puedan anidar contra la superficie de otra molécula (una interacción que subyace a la unión). El equipo de Grigoryan incluso desarrolló una red de difusión llamada Chroma y luego la acondicionó para hacer proteínas con formas que se asemejaran a las 26 letras mayúsculas que se usan en inglés, así como a los números arábigos 3 .

Señal de ruido
La pantalla de la computadora de Juergens inicialmente muestra ruido, la variedad aleatoria de aminoácidos con los que comienza el sistema de IA. Se representan como garabatos rojos y borrosos que se asemejan a la pintura con los dedos de un niño pequeño. Se transforman, fotograma a fotograma, en formas cada vez más complejas, con características similares a las proteínas, como espirales apretadas conocidas como hélices α y formas de cintas que se doblan sobre sí mismas, llamadas hojas β. “Es una buena topología mixta alfa-beta”, dice Juergens, sonriendo mientras admira una creación que tomó solo unos minutos para hacer. «Esto se ve bien».
La herramienta ha ganado un uso generalizado en el laboratorio de Baker. “El proceso de diseño es casi irreconocible en comparación con hace un año”, dice. La red neuronal se ha destacado en desafíos de diseño que han sido ineficientes, difíciles o imposibles con otros enfoques.
En un análisis informado en su estudio 1 , los investigadores comenzaron con un fragmento de otra proteína, como una porción de una proteína viral reconocida por las células inmunitarias, y asignaron herramientas basadas en IA para producir 100 proteínas nuevas diferentes, para ver cuántas incorporaría el motivo deseado. El equipo llevó a cabo este desafío para 25 formas iniciales diferentes. Los resultados no siempre incorporaron el fragmento inicial, pero RFdiffusion produjo al menos una proteína que lo hizo para 23 de los motivos, en comparación con 15 para la alucinación y 12 para la pintura.
RFdiffusion también ha demostrado ser hábil en la fabricación de proteínas que se autoensamblan en nanopartículas complejas que podrían ser capaces de administrar medicamentos o componentes de vacunas. Los enfoques anteriores de IA 5 también pueden producir este tipo de proteína, pero Watson dice que los diseños de RFdiffusion son mucho más sofisticados.
Las redes neuronales como RFdiffusion parecen realmente brillar cuando se les asigna la tarea de diseñar proteínas que pueden adherirse a otra proteína específica. El equipo de Baker ha utilizado la red para crear proteínas que se unen fuertemente a proteínas implicadas en cánceres, enfermedades autoinmunes y otras condiciones. Un éxito aún no publicado, dice, fue diseñar aglutinantes fuertes para una molécula de señalización inmunitaria difícil de atacar llamada receptor del factor de necrosis tumoral, el objetivo de los medicamentos de anticuerpos que generan miles de millones de dólares en ingresos cada año. “Está ampliando el espacio de proteínas para las que podemos crear aglutinantes y hacer terapias significativas”, dice Watson.
Pruebas del mundo real
El equipo de Baker está produciendo tantos diseños que probar si funcionan según lo previsto se ha convertido en un serio cuello de botella. “Una persona con aprendizaje automático puede generar suficientes diseños para mantener ocupados a 100 biólogos durante meses”, afirma Kevin Yang, investigador de aprendizaje automático biomédico de Microsoft Research en Cambridge, Massachusetts, cuyo equipo ha desarrollado su propia herramienta de diseño de proteínas basada en la difusión 6 .
Pero los primeros signos sugieren que las creaciones de RFdiffusion son auténticas. En otro desafío descrito en su estudio, el equipo de Baker encargó a la herramienta diseñar proteínas que contuvieran un tramo clave de p53, una molécula de señalización hiperactiva en muchos tipos de cáncer (y un objetivo farmacológico codiciado). Cuando los investigadores hicieron 95 de los diseños del software (mediante la ingeniería de bacterias para expresar las proteínas), más de la mitad mantuvo la capacidad de p53 para unirse a su objetivo natural, MDM2. Los mejores diseños lo hicieron unas 1000 veces más fuerte que el p53 natural. Cuando los investigadores intentaron esta tarea con alucinaciones, los diseños, aunque se predijo que funcionarían, no dieron resultado en el tubo de ensayo, dice Watson.
En general, Baker dice que su equipo descubrió que entre el 10% y el 20% de los diseños de RFdiffusion se unen a su objetivo previsto lo suficientemente fuerte como para ser útiles, en comparación con menos del 1% de los métodos anteriores anteriores a la IA. (Los enfoques anteriores de aprendizaje automático no podían diseñar carpetas de manera confiable, dice Watson). El bioquímico Matthias Gloegl, un colega de la UW, dice que últimamente ha alcanzado tasas de éxito cercanas al 50 %, lo que significa que puede llevar solo una semana o dos crear diseños que funcionen, en lugar de meses. «Es realmente una locura», dice.
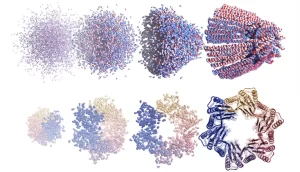
La versión basada en la nube de RFdiffusion tenía alrededor de 100 usuarios cada día a fines de junio, según Sergey Ovchinnikov, biólogo evolutivo de la Universidad de Harvard en Cambridge, Massachusetts. Joel Mackay, bioquímico de la Universidad de Sydney en Australia, ha estado incursionando en la difusión de RF para diseñar proteínas capaces de unirse a otras proteínas que estudia su laboratorio, que incluyen moléculas llamadas factores de transcripción que controlan la actividad genética en las células. Encontró que el proceso de diseño era simple y utilizó modelos informáticos para validar que, en teoría, las proteínas deberían unirse a los factores de transcripción.
Mackay ahora está probando si las proteínas pueden alterar la expresión génica según lo previsto cuando se producen en las células. Tiene los dedos cruzados, porque tal hallazgo equivaldría a una forma sencilla de activar y desactivar factores de transcripción específicos dentro de las células, en lugar de usar medicamentos que pueden llevar años identificar, si es que se pueden descubrir. “Si este método funciona de manera confiable para nuestros tipos de proteínas, sería un cambio de juego total”, dice.
Futuras mejoras
Los últimos modelos, como RFdiffusion, son un «cambio radical», dice Charlotte Deane, informática inmunológica de la Universidad de Oxford, Reino Unido. Pero quedan desafíos clave. “Lo que hará es inspirar a la gente a ver hasta dónde podemos llevar estos métodos de difusión”, dice.
Una aplicación en la que ella y otros científicos y empresas de biotecnología están especialmente interesados es el diseño de proteínas de unión más complejas, como los anticuerpos o los receptores de proteínas utilizados por las células T (un tipo de célula inmunitaria). Estas proteínas tienen bucles flexibles que se entrelazan con sus objetivos, a diferencia de las interfaces planas en forma de sándwich en las que RFdiffusion se ha destacado hasta ahora. Baker dice que están progresando con los anticuerpos.
Ovchinnikov y otros dicen que es un desafío, en general, diseñar biomoléculas cuya función dependa de regiones flexibles que les den la capacidad de adoptar muchas formas diferentes. Estas son características que han resultado difíciles de modelar usando IA. “Si el problema es, ¿podemos unirnos a algo más e inhibirlo?”, dice Ovchinnikov, “creo que ese problema se resolverá con estos métodos. Pero para hacer algo más complejo, más parecido a lo que hace la naturaleza, necesitas introducir algo de flexibilidad”.
Tanja Kortemme, bióloga computacional de la Universidad de California, San Francisco, está utilizando la difusión de RF para diseñar proteínas que pueden usarse como sensores o como interruptores para controlar las células. Ella dice que si el sitio activo de una proteína depende de la ubicación de unos pocos aminoácidos, la red de IA lo hace bien, pero tiene dificultades para diseñar proteínas con sitios activos más complejos, lo que requiere muchos más aminoácidos clave para estar en su lugar, un desafío. ella y sus colegas están tratando de abordar.
Otra limitación de los últimos métodos de difusión es su incapacidad para crear proteínas que son muy diferentes de las proteínas naturales, dice Yang. Esto se debe a que los sistemas de IA han sido entrenados solo en proteínas existentes que los científicos han caracterizado, dice, y tienden a crear proteínas que se parecen a ellas. La generación de proteínas de aspecto más extraño podría requerir una mejor comprensión de la física que imbuye a las proteínas de su función.
Eso podría facilitar el diseño de proteínas para llevar a cabo tareas para las que ninguna proteína natural ha evolucionado. “Todavía hay mucho espacio para crecer”, dice Yang.
Las últimas herramientas de diseño de proteínas han demostrado ser extremadamente poderosas en la creación de proteínas que pueden realizar una tarea particular, siempre que esa función pueda describirse en términos de una forma, como la superficie de una proteína a la que unirse, dice AlQuraishi. Pero, agrega, herramientas como la difusión de RF aún no pueden manejar otros tipos de especificaciones, como hacer una proteína que pueda llevar a cabo una reacción particular independientemente de su forma, cuando «sabes lo que quieres pero no lo sabes». saber qué es la geometría”.
Las futuras herramientas de diseño de proteínas también necesitarán la capacidad de producir proteínas según numerosos criterios diferentes, dice Grigoryan. Una proteína terapéutica potencial no solo debe unirse a su objetivo, sino que tampoco debe unirse a otras y debe poseer propiedades que faciliten su producción en masa.
Una dirección que los investigadores están explorando es si las proteínas podrían diseñarse utilizando descripciones de texto en lenguaje sencillo, similares a las indicaciones que se envían a las herramientas de generación de imágenes como Midjourney. “Realmente puedes imaginar que podremos escribir descripciones de una proteína y sintetizarlas y probarlas”, dice Watson.
Grigoryan y sus colegas han dado un paso hacia este objetivo. En su preimpresión de diciembre de 2022 3 , entrenaron a Chroma para adjuntar descripciones a sus diseños y escupir diseños con especificaciones basadas en texto, incluida ‘proteína con un dominio CHAD’ (una forma de proteína que incorpora múltiples hélices) o ‘estructura cristalina de aminotransferasas’ ( enzimas involucradas en la producción y descomposición de proteínas).
La proteína que Juergens creó en unos minutos esta mañana es solo un modelo de la estructura 3D de una proteína. Luego, Juergens usa otra herramienta de inteligencia artificial para generar secuencias de aminoácidos que deberían plegarse en esa estructura. Como verificación final, inserta las secuencias en AlphaFold para ver si el software predice estructuras plegadas que coincidan con el diseño. Son acertadas, con las predicciones de AlphaFold que difieren del diseño en un promedio de solo 1 ångström (el ancho de un átomo de hidrógeno).
“Esto tiene la precisión que clasificaríamos como un éxito de diseño”, dice Watson. Lo único que queda por hacer, dice, es ver cómo se comporta la proteína en la vida real.
Naturaleza 619 , 236-238 (2023)
doi: https://doi.org/10.1038/d41586-023-02227-y
Referencias



