Un equipo de nanobiotecnólogos del Instituto Wyss de Ingeniería Biológicamente Inspirada de Harvard y el Instituto del Cáncer Dana-Farber (DFCI) dirigido por William Shih, Ph.D., miembro de la Facultad Fundadora de Wyss, ha ideado una estrategia de autoensamblaje de ADN programable que resuelve la clave
El desafío del control de nucleación robusto y allana el camino para aplicaciones como la detección de biomarcadores de diagnóstico ultrasensibles y la fabricación escalable de estructuras de tamaño micrométrico con características de tamaño nanométrico. Usando el método, llamado «polimerización entrecruzada», los investigadores pueden iniciar el tejido de nanocintas a partir de hebras individuales alargadas de ADN (denominadas «listones») mediante un evento de nucleación estrictamente dependiente de la semilla. El estudio se publica en Nature Communications.
Las nanoestructuras de ADN tienen un gran potencial para resolver diversos desafíos de diagnóstico, terapéuticos y de fabricación debido a su alta biocompatibilidad y programabilidad. Para que funcionen dispositivos de diagnóstico efectivos, por ejemplo, una nanoestructura de ADN podría necesitar responder específicamente a la presencia de una molécula objetivo activando una lectura amplificada compatible con instrumentos de bajo costo accesibles en el punto de atención o en entornos de laboratorio clínico.
La mayoría de las nanoestructuras de ADN se ensamblan utilizando una de las dos estrategias principales, cada una de las cuales tiene sus fortalezas y limitaciones. Los «origami de ADN» se forman a partir de una hebra de andamio de una sola hebra larga que se estabiliza en una configuración bidimensional o tridimensional mediante numerosas hebras de fibras más cortas. Su montaje depende estrictamente del ramal del andamio, lo que lleva a un plegado robusto de todo o nada. Aunque pueden formarse con alta pureza en una amplia gama de condiciones, su tamaño máximo es limitado. Por otro lado, los «ladrillos de ADN» pueden ensamblar estructuras mucho más grandes a partir de una multitud de hebras modulares cortas. Sin embargo, su ensamblaje requiere condiciones ambientales estrictamente controladas, puede iniciarse de manera falsa en ausencia de una semilla y produce una proporción significativa de estructuras incompletas que deben purificarse.
«La introducción del origami de ADN ha sido el avance más impactante en el campo de la nanotecnología de ADN en las últimas dos décadas. El enfoque de polimerización entrecruzada que desarrollamos en este estudio se basa en esta y otras bases para extender el autoensamblaje de ADN controlado a mucho escalas de mayor longitud «, dijo Shih, quien codirige la Iniciativa de Robótica Molecular de Wyss, y también es profesor en la Escuela de Medicina de Harvard y DFCI. «Prevemos que la polimerización entrecruzada permitirá ampliamente la formación de todo o nada de microestructuras bidimensionales y tridimensionales con características de nanoescala direccionables, autoensamblaje algorítmico y amplificación de señal de fondo cero en aplicaciones de diagnóstico que requieren una sensibilidad extrema».
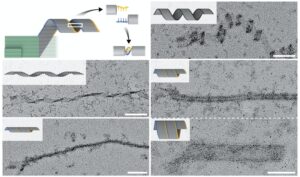
Plantar una semilla
Habiendo experimentado las limitaciones del origami de ADN y las nanoestructuras de ladrillos de ADN, el equipo comenzó preguntando si era posible combinar la dependencia absoluta de la semilla del ensamblaje de origami de ADN con el tamaño ilimitado de las construcciones de ladrillos de ADN en un tercer tipo de nanoestructura de ADN que crece rápidamente. y consistentemente a un gran tamaño.
«Argumentamos que el ensamblaje de todo o nada de estructuras de ADN a escala micrométrica podría lograrse mediante el diseño de un sistema que tenga una alta barrera de energía libre para el ensamblaje espontáneo. La barrera solo se puede sortear con una semilla que se une y organiza un conjunto de listones ‘nucleantes’ para la captura conjunta de listones de ‘crecimiento’. Esto inicia una reacción en cadena de adiciones de listones de crecimiento que da como resultado largas cintas de ADN «, dijo el coautor Dionis Minev, Ph.D., quien es becario postdoctoral en el equipo de Shih. «Este tipo de nucleación altamente cooperativa, estrictamente dependiente de la semilla, sigue algunos de los mismos principios que rigen la iniciación y el crecimiento de la actina citoesquelética o del filamento de microtúbulos en las células». El alargamiento de los filamentos citoesqueléticos sigue reglas estrictas en las que cada monómero entrante se une a varios monómeros que se han incorporado previamente al filamento polimérico y, a su vez, es necesario para la unión del siguiente. «La polimerización entrecruzada lleva esta estrategia al siguiente nivel al permitir que los vecinos no cercanos sean necesarios para el reclutamiento de los monómeros entrantes. El nivel extremo de coordinación resultante es la salsa secreta», dijo Minev.
Del concepto a la estructura (s) actual
Al poner en práctica su concepto, el equipo diseñó y validó un sistema en el que una pequeña estructura de semillas ofrece una alta concentración inicial de sitios de unión preformados en forma de hebras de ADN únicas que sobresalen. Estos pueden ser detectados por listones de ADN con seis (o en un sistema entrecruzado alternativo ocho) sitios de unión disponibles, cada uno de los cuales se une a uno de los seis (u ocho) filamentos de ssDNA vecinos que sobresalen en un patrón entrecruzado, y los listones de ADN subsiguientes se agregan continuamente a la estructura alargada.
«Nuestro diseño es notable porque logramos un rápido crecimiento de enormes estructuras de ADN, pero con un control de nucleación que es órdenes de magnitud mayor que otros enfoques. Es como tener su pastel y comérselo también, porque creamos fácilmente ensamblajes y lo hicimos sólo donde y cuando lo deseamos «, dijo el coautor Chris Wintersinger, un Ph.D. estudiante del grupo de Shih que colaboró en el proyecto con Minev. «El control que logramos con el entrecruzamiento supera con creces el observado para los métodos de ADN existentes en los que la nucleación solo puede dirigirse dentro de una estrecha ventana de condiciones en las que el crecimiento es extremadamente lento».
Utilizando la polimerización entrecruzada, el equipo de Shih generó cintas de ADN que se autoensamblaron como resultado de un solo evento de siembra específico en estructuras que medían hasta decenas de micrómetros de longitud, con una masa casi cien veces más grande que un origami de ADN típico. Además, al aprovechar la alta capacidad de programación de las conformaciones e interacciones de las lamas, los investigadores crearon cintas con giros y giros distintos, lo que resultó en estructuras en espiral y en forma de tubo. En estudios futuros, esto podría aprovecharse para crear estructuras funcionalizadas que puedan beneficiarse de compartimentos separados espacialmente. «Una aplicación inmediata de nuestro método de nanoconstrucción entrecruzada es como una estrategia de amplificación en ensayos de diagnóstico después de la formación de nano semillas a partir de biomarcadores específicos y raros», dijo la coautora Anastasia Ershova. quien también es un Ph.D. estudiante guiado por Shih.
«El desarrollo de este nuevo método de nanofabricación es un ejemplo sorprendente de cómo la Iniciativa de Robótica Molecular del Instituto Wyss sigue inspirándose en sistemas biológicos, en este caso, el crecimiento de filamentos citoesqueléticos, y sigue ampliando las posibilidades en este apasionante campo. Este avance trae la potencial de la nanotecnología del ADN más cerca de resolver desafíos de diagnóstico urgentes para los que actualmente no hay soluciones «, dijo el director fundador de Wyss, Donald Ingber, MD, Ph.D., quien también es profesor de Biología Vascular Judah Folkman en la Escuela de Medicina de Harvard y Boston Children’s. Hospital y profesor de Bioingeniería en la Escuela de Ingeniería y Ciencias Aplicadas John A. Paulson de Harvard.
Fuente: el Instituto Wyss de Ingeniería de Inspiración Biológica de Harvard . Original escrito por Benjamin Boettner.



